
Reclasificación identifica nueva especie de Tenacibaculum en Chile
Chile: Además de clasificar y comparar las secuencias genómicas disponibles para las distintas especies de Tenacibaculum, los resultados de un reciente estudio publicado en la web realizado en conjunto por investigadores de Francia, Noruega y Chile, corrige la identificación y clasificación de la cepa AYD7486TD originalmente descrita como T. dicentrarchi realizada por el laboratorio ADL Diagnostic Chile en el 2016.
El estudio “Comparative genomics of Tenacibaculum dicentrarchi and “Tenacibaculum finnmarkense” highlights intricate evolution of fish-pathogenic species” publicado recientemente en la revista Genome Biology and Evolution, presenta una comparación genómica realizada con varias cepas de Tenacibaculum incluyendo aislados de campo en Chile y Noruega. El trabajo fue realizado por un equipo multidisciplinario de Francia, Noruega y en Chile el Dr. Rubén Avendaño-Herrera de la Universidad Andrés Bello y el Centro Fondap Incar.
Lea también: Entendiendo la reclasificación de la cepa chilena AY7486TD de Tenacibaculum
Mediante la técnica de Identidad Promedio de Nucleótidos (ANI por sus siglas en inglés), los investigadores fueron capaces de definir los límites que clasifican las especies y también inferir las relaciones filogenéticas entre las cepas.
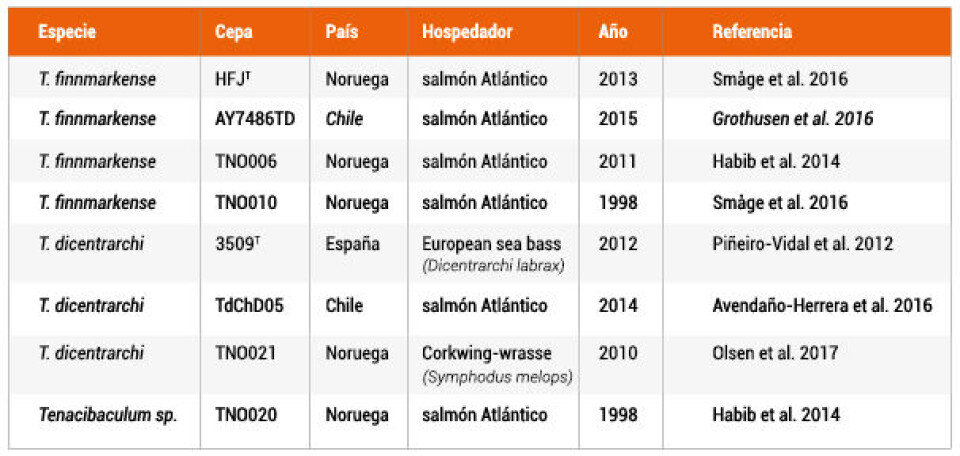
Los autores descubrieron que las cepas TdChD05 de Chile y TNO021 de Noruega, ambas previamente descritas como T. dicentrarchi, forman un grupo altamente cohesionado con T. dicentrarchi USC 3509T (cepa tipo).
Lea también: Publican primera identificación y caracterización de Tenacibaculum dicentrarchi
Por el contrario, la cepa AY7486TD, también descrita originalmente como T. dicentrarchi y publicado por ADL Diagnostic, no cae dentro del grupo de T. dicentrarchi; en cambio, forma un clúster con las cepas TNO006, TNO010 y "T. finnmarkense "HFJT, lo que denota un error en su primera clasificación.
Lea también: ADL secuenció genoma completo de Tenacibaculum dicentrarchi
Los valores ANI obtenidos por los investigadores también describen dos subgrupos en esta especie, una agrupación de las cepas TNO010 y AYD7486TD y otra agrupación de las cepas TNO006 y "T. finnmarkense "HFJT.
Los científicos exponen que a pesar de que T. dicentrarchi y "T. finnmarkense son especies distintas, “obviamente muestran proximidad significativa en términos de identidad de secuencias y fracción de genomas compartidos”.
Finalmente, encontraron que la cepa TNO020 no pertenece a ninguno de los conglomerados previamente definidos y, por lo tanto, es probable que pertenezca a una especie de Tenacibaculum aún no descrita como se sugirió anteriormente.
Lea también: Identifican y caracterizan aislados virulentos de Tenacibaculum dicentrarchi.
Al respecto, el Dr. Rubén Avendaño explicó a Salmonexpert las implicancias de este descubrimiento.
¿Qué relevancia tiene para el país la descripción de esta nueva especie de Tenacibaculum?
Es muy relevante porque demuestra la presencia de una diversidad de bacterias filamentosas del género y que son reconocidas como patógenas en el salmón Atlántico cultivado en Noruega. Además, provoca infecciones ulcerosas externas en los peces por lo que tenemos que poner mucho cuidado, ya que ahora no sólo es T. dicentrarchi, sino también otros miembros del género Tenacibaculum.
¿Ya que el aislado AY7486TD está asociado a brotes con mortalidad en el año 2015, puede que efectivamente sea más patogénica, o que se sabe al respecto?
Nuestros estudios denotan su presencia los años posteriores y asociados a cuadros infecciosos clínicos. Es muy relevante señalar que los últimos tres años se han incrementado los diagnósticos asociados o informados por los laboratorios de servicio como Tenacibaculum spp. Hoy con nuestro estudio se demuestra que existe una mayor diversidad microbiana y estamos trabajando en determinar su potencial patogénico, lo cual no debería ser distinto a otros miembros del género.
¿Cómo cree que es la distribución espacial de las distintas especies de Tenacibaculum? ¿Se encuentran ampliamente distribuidas? ¿Existe mayor prevalencia de una que de otra?
Es una gran pregunta, ya que se requieren estudios de este tipo con el fin de buscar medidas preventivas, pues este grupo de bacterias llegó para quedarse y hoy se habla cada vez mas de Tenacibaculosis en la industria del salmón. Nosotros tenemos una historia de muchos años trabajando con bacterias de este género y hoy tenemos numerosos estudios que prontamente deben ser publicados, aportando no sólo a la generación de conocimiento sino medidas de control y prevención.
¿En palabras simples, cómo se explica la relación genética entre T. dicentrarchi y T. finnmarkense?
La relación taxonómica y evolutiva de las bacterias del género Tenacibaculum recién se esta aclarando y así se puede verificar a nivel taxonómico, en donde hasta el año 2002 se conocían unas pocas especies y todas ellas proveniente del género Flexibacter y con potencial patogénico para especies de peces marinos. En los últimos años se ha incrementado la descripción de estos microorganismos, los que son componentes importantes de los ambientes acuáticos y microbiota del salmón del Atlántico. Por tanto, el desarrollo de los medios de cultivos y herramientas moleculares ha permitido abrir los ojos y observar que Tenacibaculum es un componente relevante, mas aún si siempre esta presente en cuadros infecciosos de peces cultivados.
Lea también: Investigadores chilenos estandarizan técnica para detección de Tenacibaculum dicentrarchi.
¿Cómo funcionan los métodos ANI y core genome-based phylogeny utilizados en su estudio? ¿Porqué son mas confiables que un PCR? ¿Se debería descartar la PCR como método de clasificación taxonómica para esta especie?
El ANI es una herramienta que permite comparar la relación genética entre distintas bacterias, por lo que en la actualidad es imprescindible de realizar en los estudios de identificación taxonómica de bacterias. Nuestro equipo de investigación tiene una amplia experiencia en la descripción de nuevas especies de bacterias y antes se consideraba como una herramienta valida la hibridación del ADN de una hebra de una bacteria versus la complementaria de la hebra de DNA de la otra bacteria. Esperando un valor de corte de 70% para delinear la existencia o no de una nueva especie. Hoy se considera que el 70% corresponde a un 95% del ANI y se compara todo el genoma, por lo que su robustez es mucho mayor. Por tanto, el PCR y el ANI tienen distinto fin, uno como método de diagnóstico y el otro como taxonómico. Sin embargo, mi experiencia en diagnóstico me ha enseñado que hay ciertos pasos mínimos que se deben realizar antes de realizar un diagnóstico, lo cual hoy se ha olvidado y confiamos demasiado en el PCR para decidir si la causa de muerte de los peces es una u otra. En este sentido, hay que volver al pasado y considerar como muy relevante el cuadro clínico de los peces y la observación macroscópica de los signos clínico de los peces, el intento del aislamiento del agente para identificar, caracterizar y realizar los estudios de susceptibilidad a antibióticos y en una última etapa el PCR para confirmación. Sin embargo, lamentablemente esto no ocurre y lo que es mas complejo es que en el caso de Tenacibaculum, se están empleando métodos moleculares que no están validados con un consistente número de microorganismos, por lo que la especificidad podría ser un problema.
¿Por qué cree que se produjo esta confusión en la clasificación previa del aislado AY7486TD?
Hace un par de años atrás, se generaron numerosas revistas que no tienen un Comité Editorial que se preocupe cuidadosamente de revisar lo que se publica y a mi juicio eso es lo que ocurrió.
Cuando nosotros publicamos en esta revista, estamos seguro que lo que estamos publicando es parte de un estudio completo, pero en el caso de la secuencia AY7486TD no se realizaron más estudios. De hecho, nosotros con los colegas de Francia venimos trabajando de hace mucho tiempo y escribimos a la revista que la secuencia depositada con AY7486TD no pertenecía a T. dicentrarchi, por lo que debería cambiarse a Tenacibaculum spp., pero la respuesta no fue acorde a lo que se esperaba. Si se hubiera considerado lo antes señalado, quizás la identificación de T. finnmarkense podría haberse realizado primero en Chile y por qué no haberse llamado "Tenacibaculum chilensis". Hoy estamos claros que aunque T. finnmarkense está en proceso de validación como especie, ésta existe y lo bueno es que todos los que trabajamos en estas bacterias filamentosas estamos colaborando, por tanto, tenemos un dialogo común y un interés común.
Lea el artículo completo aquí




















