PCR-RFLP: detección de Piscirickettsia en cinco horas y a bajo costo
Chile: El método diagnóstico desarrollado por chilenos permite la diferenciación de genogrupos, es de fácil implementación, entrega resultados en cinco horas y no requiere de personal altamente entrenado en biología molecular ni equipos de alto costo.
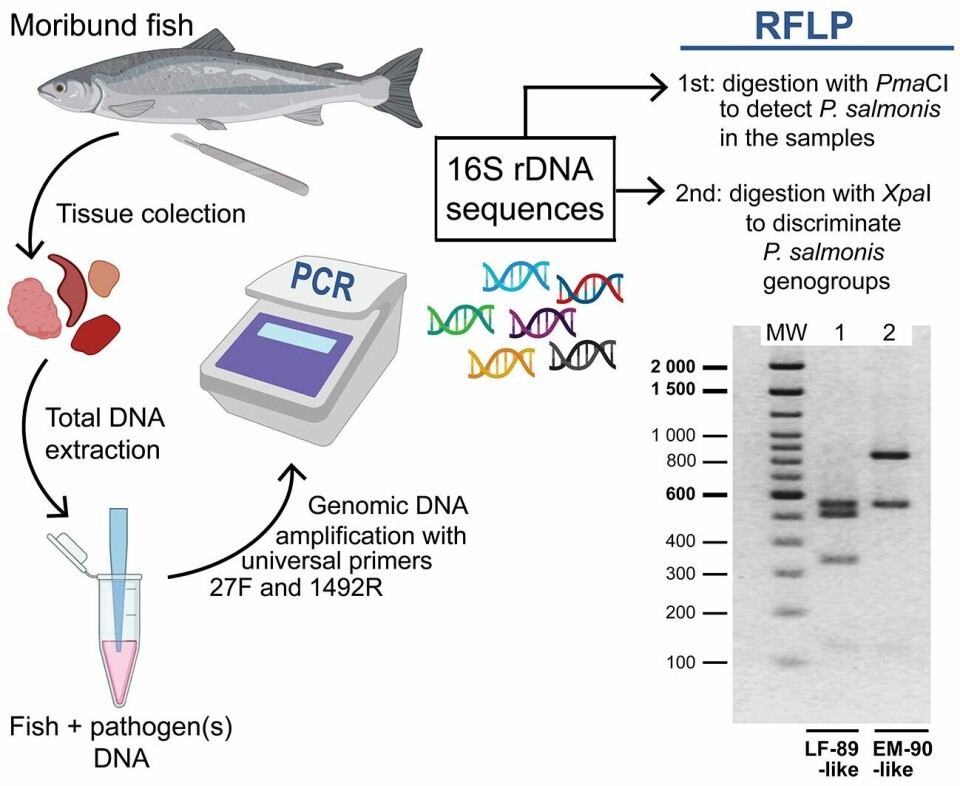
Científicos chilenos desarrollaron un PCR-RFLP (Polimorfismos de longitud de fragmentos de restricción) que permite la amplificación del gen ARNr 16S de ambos genogrupos de Piscirickettsia salmonis, y otras bacterias, para luego lograr la diferenciación de las especies mediante enzimas de restricción.
“Si bien se habían descrito algunas metodologías para diferenciar los genogrupos de P. salmonis, todas ellas eran difíciles de implementar en laboratorio, o eran demandantes en tiempo y recursos”, explica a Salmonexpert sobre el origen del desarrollo, Pamela Aravena, investigadora del Instituto de Nutrición y Tecnología de los Alimentos (INTA).
Pamela es una de los expertos que desarrollaron el PCR-RFLP. Ella comenta que ambos genogrupos poseen diferencias que hacen clave la determinación de forma rápida y a bajo costo, entre las que se encuentran la patogenicidad, signos clínicos, susceptibilidad a antibióticos, entre otras.
Así, el año 2015 ya habían implementado una metodología que les permitía evaluar la pureza de cultivos de P. salmonis mediante la técnica del PCR-RFLP (descrita en: Mandakovic y cols., 2016), sin embargo, solo les permitía detectar la presencia y pureza de P. salmonis en los cultivos y no la clasificación de los genogrupos.

Para resolver este problema lo primero que hicieron fue determinar las enzimas de restricción comerciales que generaran un patrón diferencial entre los genogrupos y que, además, ese patrón fuese sencillo de detectar en un gel de agarosa convencional.
Fue así como seleccionaron la enzima de restricción XapI, la cual validaron en laboratorio utilizando muestras de ADN extraído de cultivos puros de P. salmonis incluyendo las cepas de referencia LF-89 y EM-90 y 13 aislados.
“Nuestro desarrollo también nos permite saber si la muestra posee solamente P. salmonis o si hay otras bacterias presentes en la muestra. En las muestras evaluadas no detectamos co-infección. Mayoritariamente encontramos que las muestras analizadas presentaban infección con P. salmonis genogrupo EM-90-like” señaló la experta del INTA.
Dentro de las ventaja de esta técnica, la especialista comenta que la presencia de la bacteria y su genogrupo pueden ser evaluados directamente en el ADN extraído de tejidos de peces infectados, lo que representa una diferencia substancial con otras estrategias, tales como la secuenciación de regiones del gen 16S ribosomal, o métodos de genotipado como la tipificación multilocus de secuencias (MLST).
Asimismo, según la investigadora del INTA, este método es de fácil implementación en laboratorio, entrega resultados en cinco horas y no requiere de personal altamente entrenado en biología molecular ni equipos de alto costo, “sólo se necesitan implementos posiblemente presente en casi todos los laboratorios de diagnóstico, como un termociclador y cámaras de electroforesis. Por otra parte, los reactivos de PCR son de bajo costo, y fáciles de obtener, lo mismo que las enzimas de restricción”.
“El cultivo de P. salmonis podría tardar hasta 14 días en algunos casos y requiere incubadores refrigerados, y espacios estériles para mantener y propagar los cultivos. Además, la observación de las colonias no indica el genogrupo ni la pureza de los cultivos obtenidos. Para laboratorios de referencia o estudios epidemiológicos, nuestra metodología permite un análisis rápido, económico e independiente de cultivo” concluye.




















