
Estudio genómico revela nueva información sobre Piscirickettsia salmonis
Chile: Doce investigadores pertenecientes a nueve instituciones científicas realizaron un estudio donde describieron y analizaron el pan-genome de P. salmonis, encontrando información relevante acerca de la clasificación genética y factores de virulencia, entre otros, que ayudarán al futuro control de la bacteria.
Desde la primera descripción de la patología y aislamiento del agente etiológico realizado en Chile el año 1989, la enfermedad ha sido reportada en numerosos países, incluyendo Irlanda, Noruega, Canadá, Estados Unidos, Escocia y Turquía.
A pesar de las estrategias actuales para su control, como la vacunación, tratamiento antibiótico y otras herramientas biotecnológicas, la Piscirickettsiosis sigue presentando brotes asociados a mortalidades; dejando en evidencia la alta patogenicidad de la bacteria y su resistencia a antibióticos.
Lea también: "Comparación de la respuesta inmune en respuesta a la infección con dos cepas de P. salmonis"
En Chile, se han identificado dos genogrupos de P. salmonis asociados con las cepas LF-89 y EM-90. A pesar de los esfuerzos para caracterizar genéticamente la bacteria, falta información funcional para evaluar las divergencias genómicas en los factores de virulencia y metabolismo entre los genogrupos.
Pan-genome
El core-genome se define como el conjunto de genes compartidos por todas las cepas de la misma especie bacteriana. A su vez, el pan-genome es el conjunto de todos los genes de una especie bacteriana, es decir, el conjunto de genes compartidos y genes exclusivos de ciertas cepas.
El estudio del pan-genome permite dilucidar información clave del patógeno contenida en sus genes como la diversidad intraespecies, adaptaciones de la bacteria y diferenciaciones en mecanismos de patogenicidad.
Para abordar esta información, científicos de diversas facultades e institutos de la Universidad Austral de Chile, Universidad Mayor, Universidad Andrés Bello, centro Fondap, Beagle Bioinformatics, laboratorio ADL Diagnostic, y Laboratorio de Bioinformática Aplicada (Brasil), realizaron un estudio in-silico con los genomas de 19 cepas de P. salmonis aisladas de distintas zonas geográficas de Chile.
Dos genogrupos
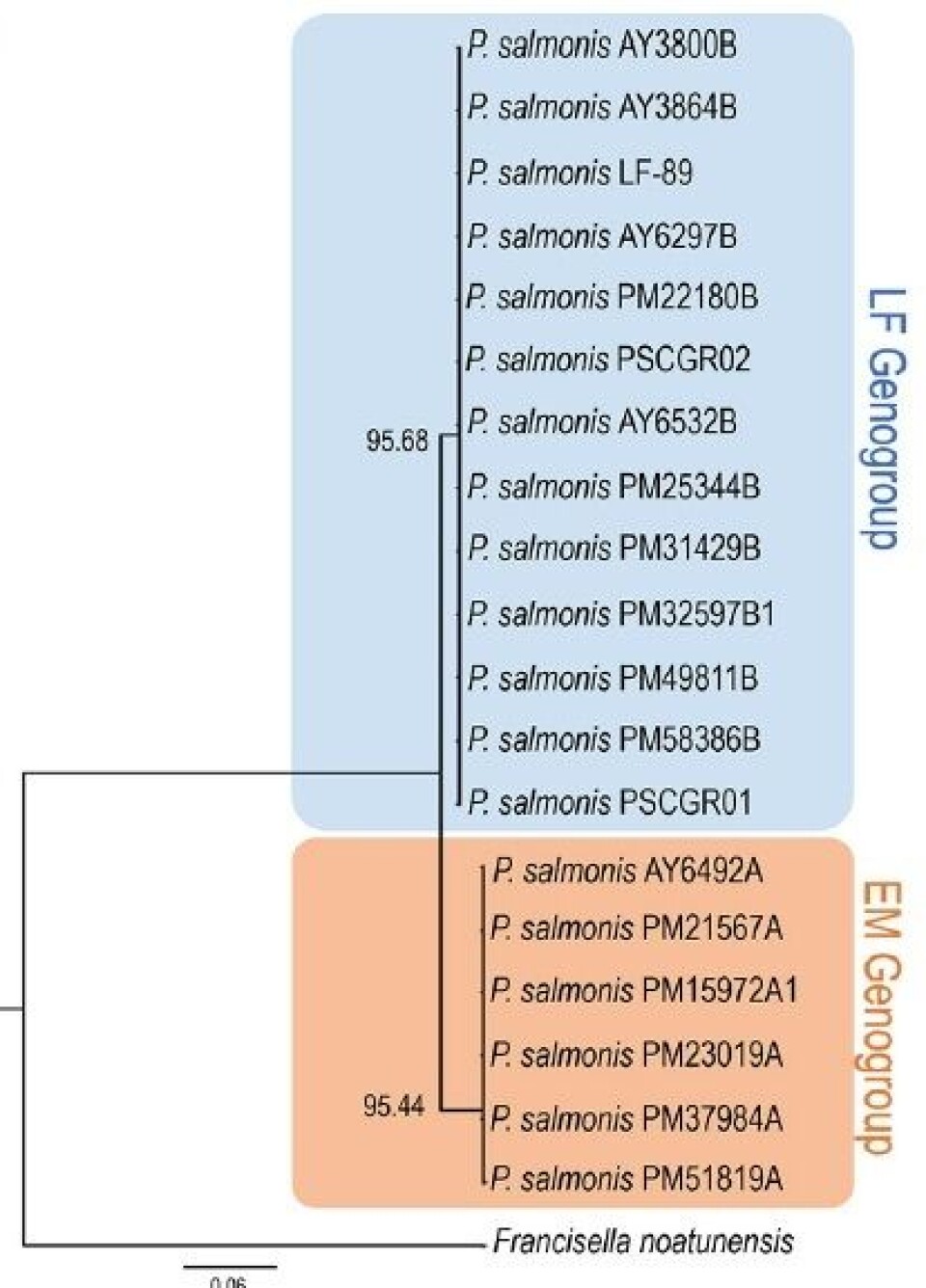
El árbol filogenético construido por los investigadores con 7 genes conservados corroboró la existencia de dos genogrupos diferentes compuestos por 6 cepas en el grupo “EM” o “A” y 13 en el grupo “LF” o “B”.
Respecto de esto, los autores informaron que: “Cada genogrupo presentó diferencias en los reordenamientos del ADN, con más divergencias observadas en los aislados del genogrupo EM. Las divergencias de este grupo incluyeron variaciones en los operones ribosomales, lo que sugiere una potencial evolución en dos nuevos subgrupos”.
Lea también: "Cepa "PS-EM-90" de P. salmonis resultaría ser más patogénica"
La confirmación de estos dos genogrupos también tiene relevancia para el desarrollo de vacunas, donde será importante mostrar si existen diferencias inmunogénicas entre los genogrupos, dado que las vacunas actuales se basan principalmente en las variantes LF.
Por otro lado, el análisis del pan-genome de P. salmonis se determinó en estado “abierto”, es decir, que posee la capacidad de adquirir ADN exógeno, lo que podría explicar la variabilidad genómica observada.
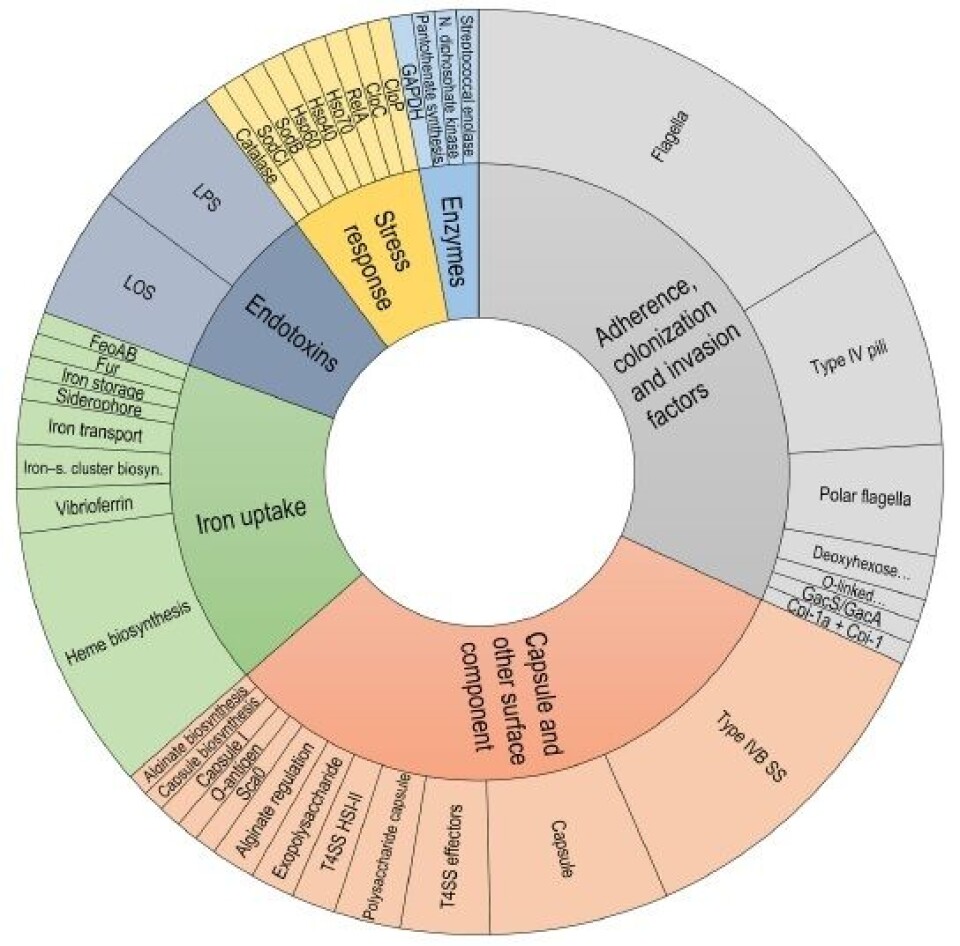
Los autores también encontraron factores de virulencia comunes para los dos grupos y los dividieron en 6 categorías: (i) endotoxinas; (ii) absorción de hierro, (iii) cápsula y otros componentes de la superficie, (iv) factores de adherencia, colonización e invasión, (v) enzimas y (vi) categoría de respuesta al estrés.
Curiosamente, algunos factores de virulencia fueron específicos de algún genogrupo, como por ejemplo, los aislados del genogrupo LF presentan una proteína de tipo “patatina”, que posee actividad fosfolipasa, asociada con la entrada de la bacteria, escape fagosómico y propagación célula a célula.
Como conclusión, los científicos manifestaron que, “los datos obtenidos sirven como un punto de partida para desarrollar metodologías innovadoras que ayuden a la discriminación entre cepas y genogrupos de P. salmonis, los que serán útiles para diagnosticar la enfermedad y para seleccionar posibles genes candidatos que podrían aplicarse en el control de la Piscirickettsiosis”.
Revise el estudio aquí.




















