Presentan resultados de estudio a microbioma de piel y branquias en infección por Piscirickettsia
Chile: Al definir el patobioma del salmón Atlántico infectado por Piscirickettsia salmonis, chilenos descubrieron que las úlceras causadas por el patógeno también eran colonizadas por otras bacterias.
La piel del salmón está cubierta por mucus que forma una capa la cual es colonizada por una compleja comunidad microbiana. La evidencia indica que las asociaciones positivas entre las bacterias asociadas a la mucosa y el sistema inmunológico del huésped protegen a los peces de la colonización por bacterias patógenas
Las branquias por su parte, también son un órgano en contacto con el medio ambiente, y son varios los factores que influyen en la composición su microbioma, como por ejemplo, el estado de salud de los peces, la nutrición y la calidad del agua.
Comprender la dinámica de las comunidades microbianas tanto dentro de la piel como de las branquias es fundamental para mejorar el rendimiento, crecimiento y bienestar de los peces.
Debido a esta razón, un nuevo estudio colaborativo realizado por científicos chilenos del Centro de Investigaciones Biológicas Aplicadas (CIBA), Universidad San Sebastián, Pontificia Universidad Católica de Chile, Universidad Católica del Norte, Universidad de Chile y la Universidad de Idaho, caracterizó y comparó la composición bacteriana de la piel y branquias de salmones sanos y de salmones cursando con una infección por Piscirickettsia salmonis.
Para ello, los investigadores utilizaron muestras de campo provenientes de centros con brotes y sin brotes de SRS en las regiones de Los Lagos y Aysén. Un total de 42 salmones Atlántico de entre 2,8 kg y 3,1 kg fueron muestreados en nueve centros de cultivo.
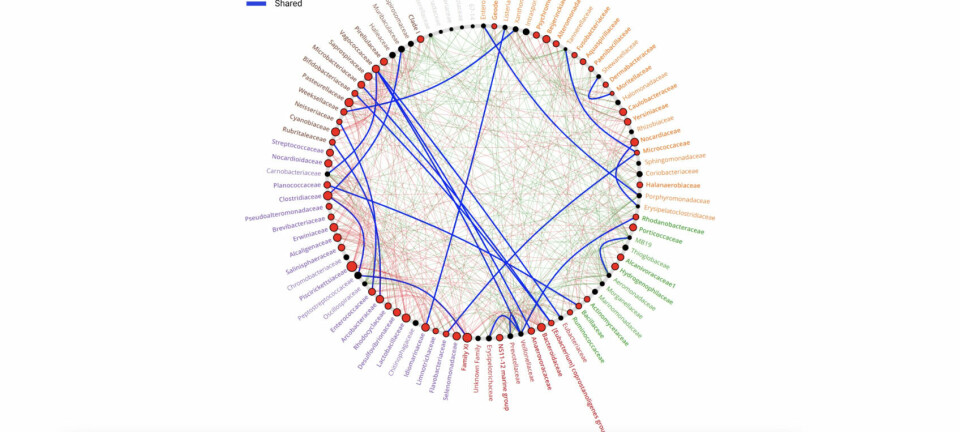
Utilizando análisis de diversidad genética en base al gen 16S ARNr, los autores encontraron diferencias significativas en los microbiomas de peces sanos y enfermos y en ambos tejidos.
“Nuestro examen del brote de úlceras en el salmón Atlántico de cultivo relacionado con P. salmonis indica la presencia de comunidades bacterianas multifacéticas, con la revelación de un patobioma central que no había sido identificado previamente. Esto subraya el potencial del análisis del ARNr 16S para discernir interacciones bacterianas intrincadas durante las infecciones”, explicaron los expertos.
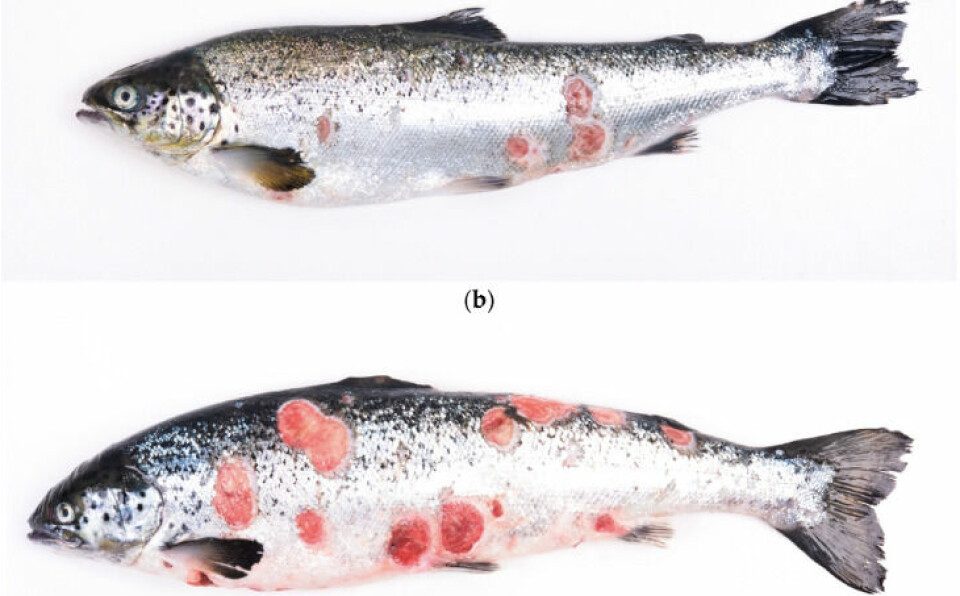
A nivel taxonómico, se observó la presencia de los órdenes Piscirickettsiales, Flavobacteriales y Pseudomonales en las muestras de piel y úlceras de peces infectados.
Luego, a nivel de especies bacterianas detectadas, los científicos descubrieron que en las úlceras fueron colonizadas principalmente por P. salmonis, Tenacibaculum dicentrarchi y Aliivibrio wodanis. “Este grupo particular de bacterias indica una estrecha correlación entre estos taxones y el estado ulcerativo observado en el salmón Atlántico. En el tejido cutáneo infectado existe una presencia significativa del género Vibrio spp. El predominio de este género en el tejido cutáneo infectado sugiere su papel potencial en las infecciones cutáneas del salmón”, señalaron los investigadores.
En branquia, a pesar de encontrar diferencia entre los microbiomas bacterianos, los autores concluyeron que no fueron significativas, sugiriendo un papel mínimo o sin impacto de P. salmonis en la alteración de la microbiota branquial en las condiciones del estudio.
A la luz de estos resultados, los científicos plantearon que comprender la dinámica de la evolución patógena en las úlceras “es fundamental para desmitificar el mecanismo de infección de P. salmonis y rastrear su vía de colonización dentro del huésped. Este descubrimiento plantea preguntas pertinentes sobre las funciones específicas de los taxones infecciosos y oportunistas detectados, en particular Vibrionaceae, Flavobacteriaceae, Piscirickettsiacea y Pseudomonadaceae, en la aparición de úlceras”.
Lea el estudio completo titulado “Salmo salar Skin and Gill Microbiome during Piscirickettsia salmonis Infection”, aquí.