Estudio confirma existencia de tres genogrupos de Piscirickettsia

Chile: En base al análisis de 73 secuencias genómicas del patógeno, tanto de Chile como de Europa y América del Norte, científicos evidenciaron la existencia de tres genogrupos distintos.
Esta evidencia fue ahora publicada oficialmente en el The ISME Jornal, de Nature, en base a los resultados de un gran estudio colaborativo realizado por científicos chilenos y alemanes.
“Aquí, dilucidamos su diversidad, evolución y selección global durante las intervenciones humanas. Nuestro análisis exhaustivo de 73 secuencias genómicas cerradas y de alta calidad cubrió cepas de brotes importantes y se complementó con un análisis de todas las secuencias del gen 16S rRNA de P. salmonis y lecturas metagenómicas disponibles en bases de datos públicas”, explican los investigadores en su publicación.
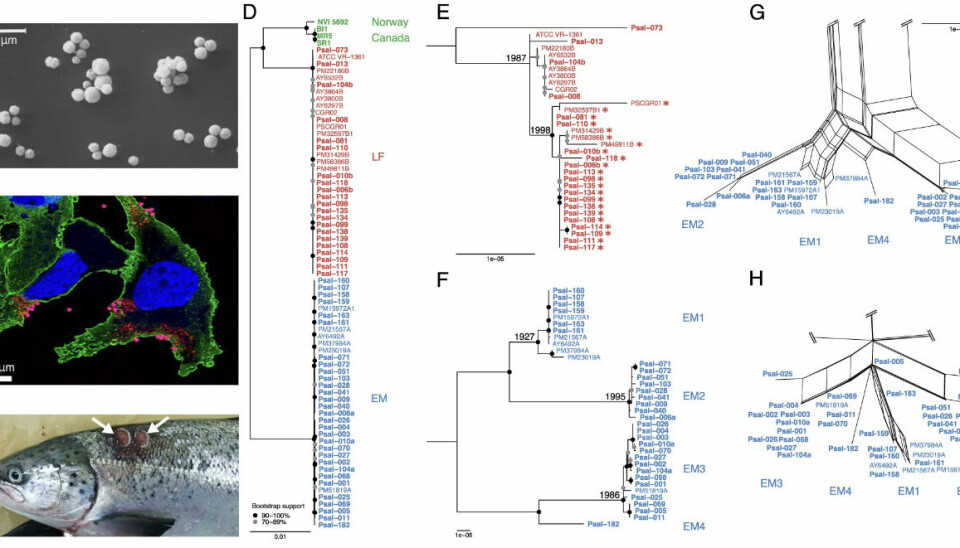
Anteriormente, se habían detectado y clasificado dos clados de P. salmonis (LF y EM) basándose en comparaciones de secuencias de genes ribosomales y de 19 genomas publicados. Sin embargo, en esta oportunidad, además de incluir 73 genomas completos, se incluyeron cepas aisladas en Europa y América del Norte, algunas de las cuales pueden representar linajes distintos.
“Nuestros 73 genomas en realidad se agruparon en tres genogrupos distintos y altamente respaldados. La mayoría de las secuencias eran miembros de los genogrupos LF y EM previamente establecidos, mientras que los cuatro aislados noruegos y canadienses formaron un genogrupo separado (NC) que está relacionado con los aislados de LF chilenos y en el que las cepas canadienses constituyen un subgrupo distinto. Lo más importante es que el presente estudio cubrió un número mucho mayor de aislamientos de EM que cualquier investigación anterior y permitió la detección de cuatro subgrupos de EM bien separados (EM1 a EM4) con un alto soporte”, detallan los expertos.
Otra de las características bacterianas evaluadas por los científicos fueron las enzimas transposasas, donde detectaron entre 331 y 848 genes de transposasa en los cromosomas de P. salmonis, lo que representa entre el 10,5 y el 21,1% de todas las secuencias codificantes, superando significativamente los porcentajes en la mayoría de otros genomas bacterianos (<3%) comparados. De acuerdo con los autores, los tres aislados canadienses tenían la mayor cantidad de genes de transposasa jamás reportados para procariotas.
“Un sello distintivo de la diversificación de P. salmonis es la cantidad y diversidad sin precedentes de transposasas que son particularmente activas en subgrupos que experimentan una rápida especiación y son clave para la adquisición de nuevos genes y para la pseudogenización”, indicaron los investigadores.
Adicionalmente, los resultados demostraron que varios genes específicos de los genogrupos están implicados en la síntesis de antígenos de superficie, los que podrían explicar las diferencias de virulencia entre cepas. “Sin embargo, el frecuente fracaso del tratamiento con antibióticos en los brotes de piscirickettsiosis no puede explicarse por la adquisición horizontal de genes de resistencia, que hasta ahora sólo se ha producido en muy raras ocasiones”, aclararon los especialistas.
Lea el estudio completo titulado “Ongoing diversification of the global fish pathogen Piscirickettsia salmonis through genetic isolation and transposition bursts”, aquí.























